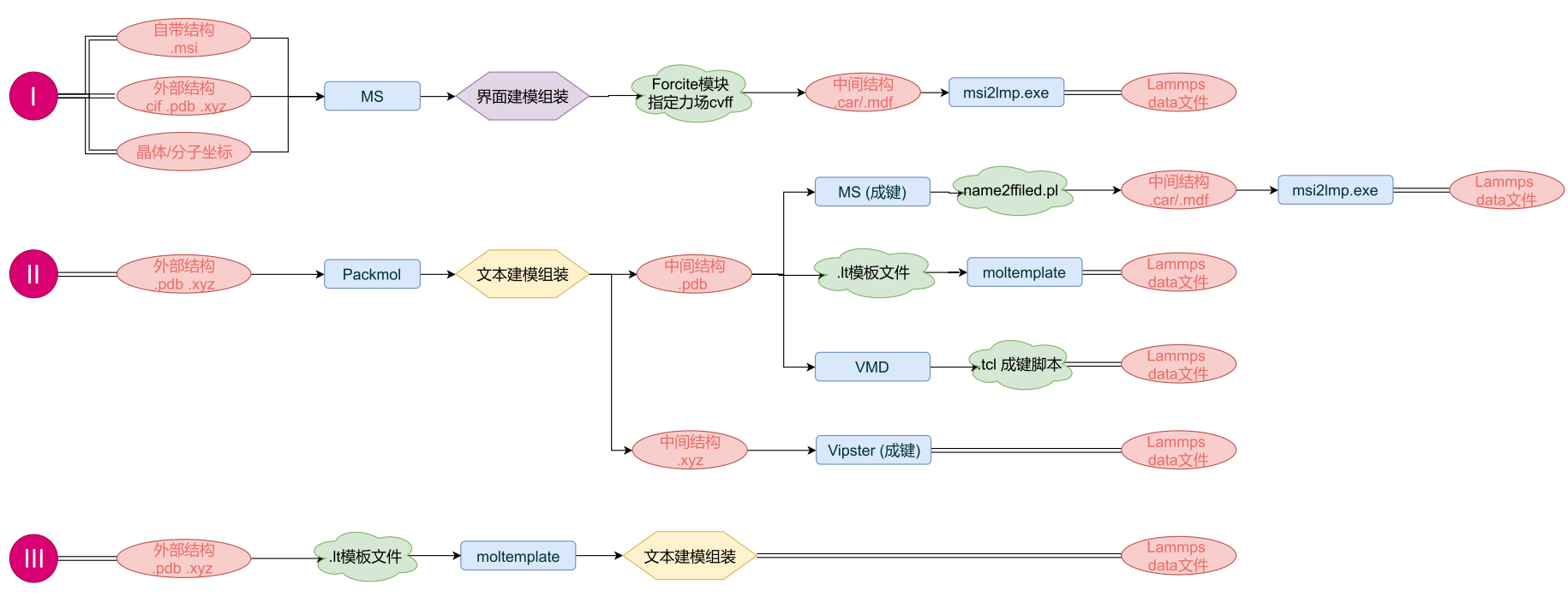
用VMD转换data文件
参见博文《VMD在LAMMPS中的应用》
moltemplate
安装
其实不用安装,因为lammps_root/tools里就有,如果需要安装,参见:http://www.moltemplate.org/download.html
使用
Example可参见:http://www.moltemplate.org/visual_examples.html
以“Coarse-grained lipid bilayer”为例:
给moltemplate准备的输入文件:
- xyz文件:system.xyz
- moltemplate文件:lipid.lt、water.lt、system.lt
具体步骤如下:
- 用packmol等生成体系的xyz文件;
- 编写lipid.lt和water.lt文件。二者分别包括了1个lipid和1个water的分子的原子参数及成键/非键相互作用参数,可以先用topotools来生成1个lipid和1个water的data文件然后在其基础上进行修改来得到。
- 编写system.lt文件。其中包括了导入lipid.lt与water.lt的信息以及对全系统的处理,如:分别导入多少个lipid分子、多少个water分子,lipid分子中原子与water分子中原子的非键相互作用参数等。
- 用moltemplate来基于system.xyz和system.lt生成
moltemplate.sh -xyz system.xyz system.lt。
pysimm:Python Simulation Interface for Molecular Modeling
基于python的lammps建模和模拟工具,主要适用于Dreiding和GAFF2力场(需要USER-MISC ,但是[这个package已经不复存在了(https://matsci.org/t/user-misc/64008)),v1.1也支持charrm力场了,看repository里也有pcff力场。建模靠一个个添加原子或.mol格式文件,聚合物里面只有PE等少数单体的,如果自己特异性的话需要单独编写然后放到monomers文件夹里。
- PySIMM tutorial paper:describes best practices of work with PySIMM and particularly discusses the construction of polymer chains with specific tacticity
- 建模及计算实例
msi2lmp
MS填充水分子
- 设置 SWNT_water.xsd为活性文件,采用 工具栏中Atom volumes and Surfaces创建一个Connolly surface。
- 等值面描述的是原子被占据的体积,如果尝试在此等值面上填充,将会发现计算终止为一个错误:" isosurface enclosed volume中的密度比要求的密度大"为了使得在 connollysurface填充,需要插入等值面的定义。可以采用 Display Style对话框达到目的。
- 选择等值面,右击显示栏,选择 Display Style,打开 Display Style对话框。在 Isosurface栏中,选中 High values inside然后关闭对话框。
- 下面可以在等值面内填充。
- 在 Amorphous Cell Calculation对话框,点击 Task中的 More…按钮打开 Amorphous Cell Packing对话框,选择 Pack in isosurface enclosed volume,然后关闭对话框。点击 Run按钮。
- 当计算结束后,可以检查密度和载荷。打开 SWNT_water.txt文件。将会发现水分子的载荷为 558个水分子。
- 将SWNT_water.xtd活动文档,并在Properties Explorer中将Filter更改为Symmetry System。在 Properties Explorer中,将 Filter改为 Symmetry。单胞的总密度设置为 1.2 g cm-3。
-
选择 File Save Project然后点击 Windows Close All。
参考资料:将分子填充到现有的结构中
参考:
- 个人总结导出data的方法
- msi2lmp的使用方法和晶体结构转化详解
- LAMMPS data文件创建工具–moltemplate
- lammps和gromacs聚合物建模的方法
- Lammps data文件建模格式转换进阶:II
手机版“神探玺洛克”请扫码